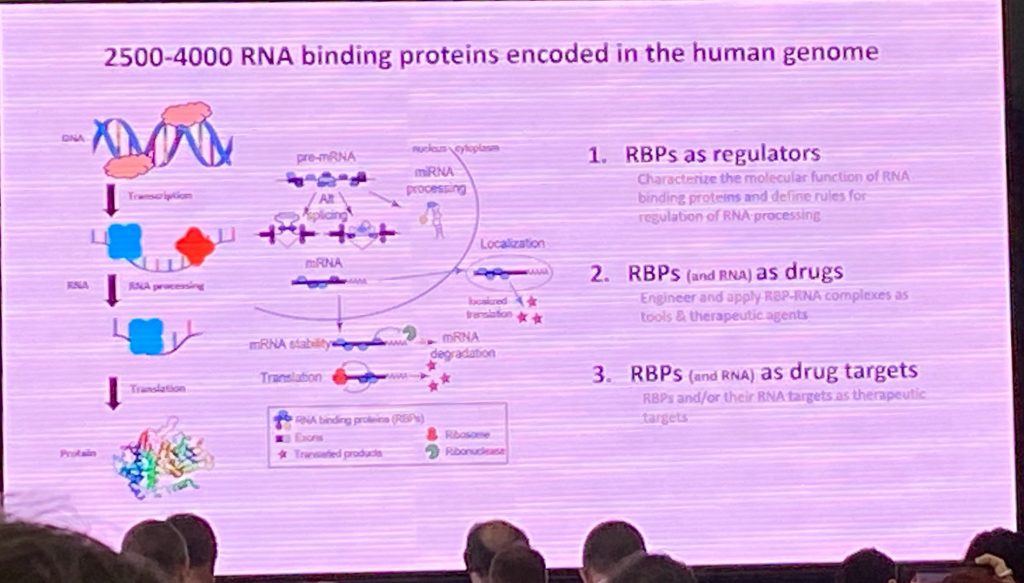
Dr. Gene Yeo da UCSD falou sobre seu trabalho recente no direcionamento da degradação do RNA usando Crispr-Cas13d. Seu laboratório se concentrou em proteínas de ligação ao RNA (RBPs) como reguladores da expressão gênica e como alvos de drogas. Ele desenvolveu métodos para estudar sistematicamente as funções RBP, como o método eCLIP aprimorado, que seu laboratório fez para caracterizar mais de 200 RBPs. Ele também desenvolveu métodos para rastrear o RNA usando imagens ao vivo em células individuais. Seus métodos foram usados para direcionar RNAs de expansão repetida usando a tecnologia Crispr-Cas. Recentemente, ele se concentrou no uso da enzima Cas13d para degradar RNAs específicos, incluindo HTT, visando a repetição CAG. O sistema Cas13d/cripsr pode caber em AAVs e, portanto, pode ser usado para estudos in vivo sem ter que usar um sistema de vetor duplo. Ele mostrou que esse sistema pode degradar o RNA do HTT e corrigir algumas das alterações moleculares encontradas nas iPSCs HD, em uma colaboração entre seu laboratório e o laboratório da Dra. Leslie Thompson.
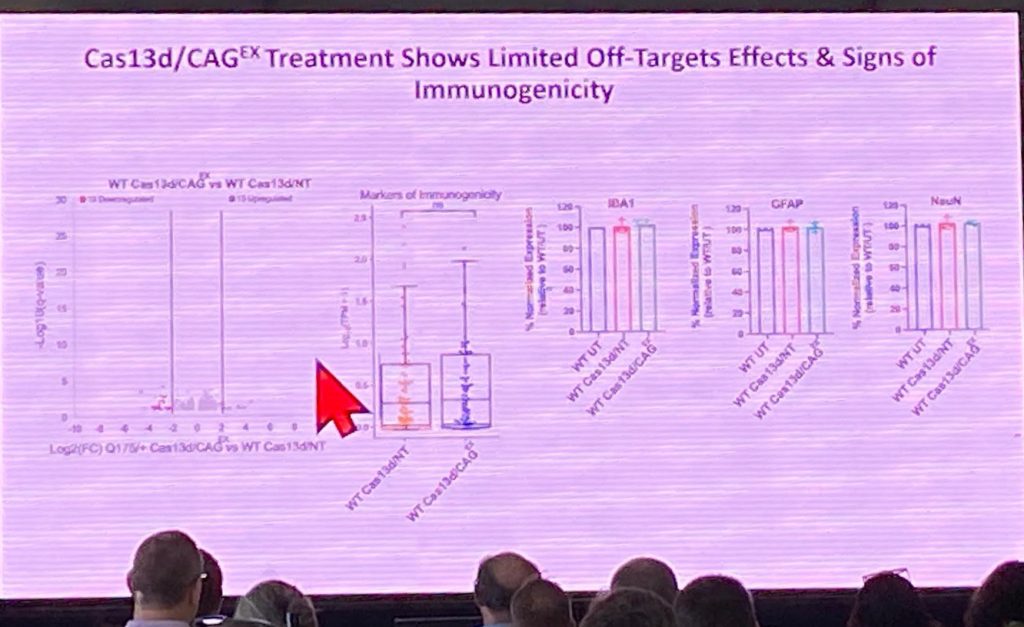
Gene passou a descrever que seu sistema tem menos efeitos fora do alvo em outros genes repetidos contendo CAG. Experimentos com roedores com AAV-Cas13d/CAGEx em Q175s podem melhorar os efeitos comportamentais neste modelo, diminuir a patologia agregada HTT e também restaurar o volume estriatal. O efeito no Q175s parecia ser específico do alelo, já que nenhuma alteração no HTT de camundongo selvagem foi detectada por Western blots, sem evidência de inflamação ou efeitos fora do alvo. Seu laboratório agora está se concentrando em outros RBPs humanos para melhorar o desenvolvimento terapêutico humano. Análise adicional significativa está faltando em minha opinião em termos de especificidade da abordagem e sua aplicabilidade para o desenvolvimento terapêutico, mas esta abordagem é significativa na medida em que visa o RNA em vez do DNA usando crispr (portanto, não editando permanentemente o genoma, incluindo locais), permitindo potencial segmentação específica do alelo que pode ser melhor tolerada.
Dra. Irina Antonijevic da Triplet Therapeutics descreveram seu trabalho no desenvolvimento de um oligonucleotídeo terapêutico antisense (ASO) direcionado ao gene Msh3, um gene crítico que controla a expansão somática da repetição HTT CAG no cérebro, conforme identificado por estudos de GWA (associação ampla do genoma). Inicialmente, o Msh3 demonstrou regular a instabilidade somática do gene HTT em camundongos, e os estudos de associação genética humana apoiaram a conservação da função do Msh3 no controle da taxa de expansão da repetição CAG e da idade de início do diagnóstico motor em indivíduos com DH.
O SHIELD-HD é um estudo de história natural conduzido pela Triplet que incluiu 70 indivíduos em 9 locais em 5 países (EUA, Canadá, França, Alemanha e Reino Unido). O estudo terá uma duração de 2 anos e apoiará o estudo planejado de Fase 1/2a com a ASO. Irina descreveu os dados iniciais sobre as alterações estruturais da ressonância magnética no volume caudado e no volume ventricular durante o estudo longitudinal durante um período de 48 semanas, sugerindo que a imagem pode ser adequada como um ponto final de biomarcador a ser rastreado. As medições do PIN HD incluem uma composição da pontuação motora total, SDMT (teste de modalidade de dígito único) e pontuações do avaliador, e também mostraram mudanças ao longo do período monitorado até o momento. Eles também estão medindo os níveis de NFL (cadeia leve de neurofilamentos) no LCR, mas ela não relatou resultados.

Irina então descreveu o trabalho recente em NHPs, o trabalho feito para promover o TTX-3360, o candidato clínico ASO. A extensão da supressão de Msh3 após a administração de ICV é grande e sustentada por um período de 24 semanas (>50% supressão é o alvo para Msh3, com base no trabalho em roedores sugerindo que esse nível de supressão deve modular SI da repetição HTT CAG). Finalmente, eles descreveram o trabalho de biomarcador feito em TTX-3360 usando medições no LCR de mRNA Msh3 contido dentro de exossomos derivados do cérebro (vesículas extracelulares). As amostras SHIELD-HD de CSF foram essenciais para o desenvolvimento do ensaio para detectar os níveis de Msh3 e alterações após a administração de ASO. A plataforma final incluiu análise qPCR para detecção de mRNA em LCR humano e primata. Os genes de controle doméstico incluem HKG, ActB e EEF2 para normalização das alterações no CSF após os tratamentos. O uso desses genes de controle foi corroborado com as amostras de LCR do estudo SHIELD-HD, garantindo que não houve alteração em indivíduos não tratados ao longo do tempo. Nos estudos de NHP, a coleta de LCR na cisterna magna foi melhor para a detecção de Msh3 do que as coletas via coleta intratecal (lombar) de LCR. Isso está pendente de confirmação em estudos de coleção humana. Eles também mediram a expressão de Msh3 saudável e HD em vesículas de LCR, que não mudaram com a doença (mas o tamanho da amostra foi pequeno). Existe uma variabilidade de 2/3 vezes na expressão entre os indivíduos medidos. Nenhum dado longitudinal foi analisado ainda, embora esta seja uma área crítica de trabalho.
Aliás, eles relatam que a distribuição de tamanho das vesículas extracelulares isoladas do LCR muda no curso da doença – então esse é um aspecto que eles querem aprofundar. Nenhum dado foi apresentado ainda in vivo, de que a dosagem de ASO altera os níveis de Msh3 em EVs do LCR que se correlacionam com a redução do parênquima do alvo. Este é o estudo crítico que está pendente.




